Hieff NGS® OnePot II DNA Library Prep Kit for Illumina/ Illumina 酶切DNA建库试剂盒
产品介绍
Hieff NGS® OnePot II DNA Library Prep Kit for Illumina®是针对Illumina®高通量测序平台专业开发设计的新一代酶切法建库试剂盒。与传统的建库法比较,本品采用高质量的片段化酶,摆脱了繁琐的超声过程,同时简化了操作流程,将片段化模块与末端修复模块合二为一,极大的降低了建库的时间和成本。本试剂盒具有优秀的文库转化率,可应用于常规动植物基因组、微生物基因组等样本,同时能兼容FFPE DNA样本的建库。经测序验证,不同GC含量的样本,均可获得优异的测序结果,文库覆盖度高,均一性好,偏好性低,使建库变得更加简单高效。
产品特色
- 适用500 pg-1 μg的基因组DNA、全长cDNA等样本。
- 高质量片段化酶,可随机切割双链DNA,酶切片段无偏好性。
- 片段化、末端修复/加A一步完成。
- 强扩增效率的高保真酶,显著提高文库质量及产量。
- 适用于FFPE DNA样本。
- 严格的批次性能与稳定性质控。
应用案例
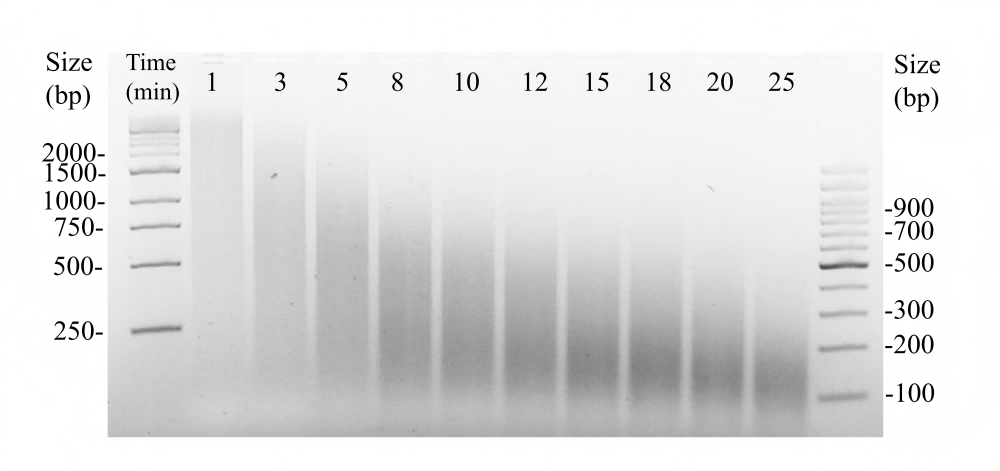
OnePotII DNA 建库试剂盒酶切 800 ng 小牛胸腺 gDNA 样本(不同酶切时间片段化效果检测)
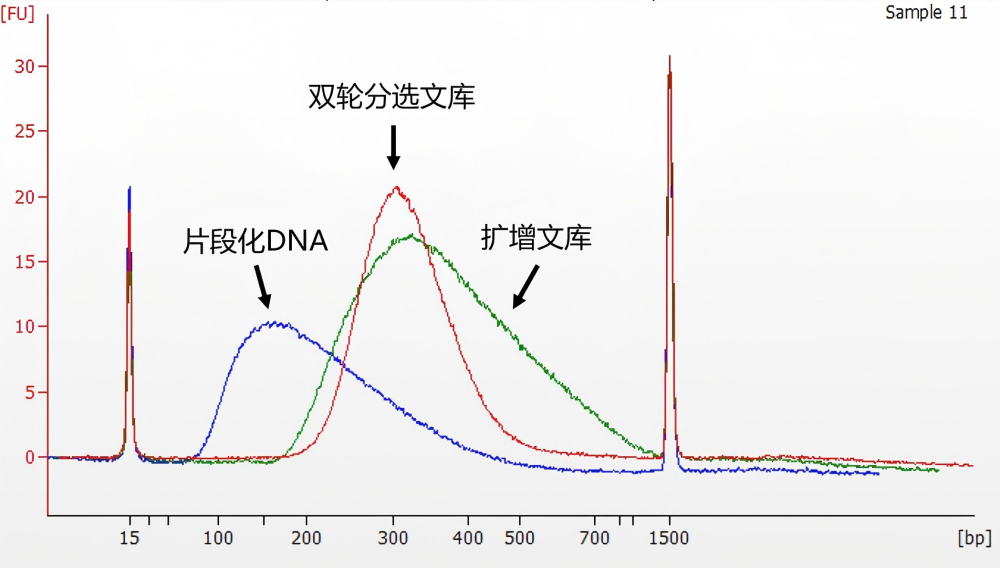
使用本试剂盒进行 human gDNA 样本建库,文库进行电泳检测(Input DNA 为 500 ng,酶切 20 min,扩增 5 cycles)
存储条件
-25~-15℃保存。
COA
已发表文献
[1] Liu K, Deng S, Ye C, et al. Mapping single-cell-resolution cell phylogeny reveals cell population dynamics during organ development. Nat Methods. 2021;18(12):1506-1514. doi:10.1038/s41592-021-01325-x(IF:47.990)
联系我们